Atualização do Comparador Biométrico com Rede Neural Siamesa: Busca 1:N
 Compartilhar no LinkedIn
Compartilhar no LinkedIn
Dando continuidade ao desenvolvimento do protótipo de um comparador biométrico funcional, implementei uma melhoria significativa: agora é possível realizar buscas do tipo 1:N, utilizando a mesma rede siamesa já treinada.
Essa evolução amplia consideravelmente a aplicabilidade do sistema, mantendo as premissas fundamentais do projeto:
- ✅ Baixo custo computacional
- ✅ Alta escalabilidade
- ✅ Execução viável em ambientes locais com infraestrutura limitada
- ✅ Possibilidade de utilização também em infraestrutura offline, ideal para ambientes sensíveis ou isolados para identificação de grupos predefinidos de pessoas.

🧠 Relembrando o conceito do Projeto
Como mencionei no post anterior, um dos objetivos deste projeto é simplificar o pipeline de verificação biométrica, ❌evitando ou otimizando etapas clássicas presentes em sistemas tradicionais, como:
- Segmentação da imagem
- Normalização via histograma
- Extração de minúcias por regras heurísticas
- Matching geométrico com validação espacial
🚀 O Que Mudou
A principal evolução foi a incorporação de uma base vetorial de embeddings, o que habilita buscas 1:N de forma simples e rápida. Para isso, criei um script específico chamado create_database.py, responsável por:
- Carregar o modelo siamesa treinado (em formato
.h5) - Aplicar o pré-processamento padronizado nas imagens da base (ex: conversão para escala de cinza, redimensionamento e normalização)
- Utilizar a sub-rede geradora de embeddings (
base_model) para codificar cada imagem como um vetor latente - Armazenar os vetores resultantes (
templates.npy) e os nomes de arquivos correspondentes (filenames.txt)
Outras alterações foram aplicadas ao app.py para gerenciamento de respostas a partir Flask.
🔄 A Nova Abordagem
O modelo proposto substitui as etapas convencionais por uma arquitetura mais enxuta e eficiente:
- Pré-processamento direto para normalização da entrada
- CNN Siamesa que gera embeddings latentes ao invés de extrair minúcias manualmente
- Distância Euclidiana entre vetores como métrica de similaridade, eliminando matching geométrico
Com isso, conseguimos um sistema mais robusto, contínuo, e adaptado a diversas condições de entrada, sem sacrificar desempenho.
🚀 Como resultado, esperamos:
- Inferência e treinamento possíveis com CPUs convencionais, sem necessidade de GPUs;
- Viabilidade de operação em:
- Ambientes locais (on-premises)
- Servidores portáteis ou embarcados
- Infraestruturas com orçamento restrito
- Execução local completa: desde a captura até a verificação, tanto para uso operacional quanto para desenvolvimento, ensino ou pesquisa.
Para isso, a abordagem que estou consolidando é baseada em:
- Redes siamesas convolucionais (CNN) que extraem embeddings discriminativos;
- Pipeline vetorial contínuo, onde cada impressão é mapeada para um vetor latente fixo;
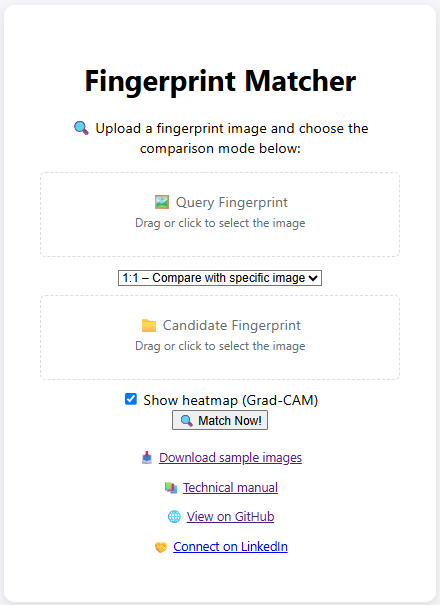
📂 Repositório + Demo
🔗 Demo operacional: https://projetos.tiago.cafe/
🔗 GitHub: https://github.com/algodas/BiomatchML.git
Member discussion